Unitea とフリーの pacs である dcm4chee で内視鏡画像システムを構築することになったのですが、それだけの設定であればとても簡単です。
でも根本的な問いかけとして、そもそも内視鏡画像を dicom 化する必要はあるのかという疑問があります。
内視鏡画像って、CTのように輝度やら何やらをあまりいじることはありません。もちろん計測はできません。ほとんどの人はほとんどそのまま閲覧しているはずです。
だったら jpg でもいいじゃないかということになりませんか?
もちろん、Unitea と接続した dcm4chee でも内視鏡画像は閲覧できますが、jpg でも閲覧できるようにしたい。
その理由は、dicom ファイルを jpg に変換する場合に、dicom ファイルのタグ情報を読み込むことになり、そうであればタグ情報を元にしたデータベースを作成し、そこにいろいろくっつけるととても便利になるからです。
pacsdb のデータベースから dicom ファイルを抽出する
これ、以前もやったのですが、別のマシンから pacs のデータベースに入って、dicom ファイルの保存場所をつきとめます。
例えば、以下のようなクエリを発行すると、カルテ番号「111111」の患者さんの dicom ファイルの保存箇所が特定できます。
SELECT filepath FROM files
WHERE instance_fk IN
(SELECT pk FROM instance WHERE series_fk IN
(SELECT pk FROM series WHERE study_fk IN
(SELECT pk FROM study WHERE patient_fk IN
(SELECT pk FROM patient WHERE pat_id = '111111'))));
python では、
def selectDcm( self, karteNo ):
conn = pymysql.connect(host=self.dbhost,
user=self.dbuser,
db=self.dbname,
password=self.dbpass,
cursorclass=pymysql.cursors.DictCursor)
try:
with conn.cursor() as cursor:
sql = "SELECT filepath FROM files WHERE instance_fk IN (SELECT pk FROM instance WHERE series_fk IN (SELECT pk FROM series WHERE study_fk IN (SELECT pk FROM study WHERE patient_fk IN (SELECT pk FROM patient WHERE pat_id = %s))));"
param = ( karteNo, )
cursor.execute( sql, param )
self.dcmfnarr = cursor.fetchall()
conn.commit()
finally:
conn.close()
で、self.dcmfnarr の中にファイルパスが格納されます。
これをローカルにダウンロードして処理します。
やっぱり ftp でダウンロード
scp の方が簡単だと思いますが、ftp の方が遥かに速いです。
約 4 倍ほど速い。
イントラネットなので ftp にします。
def dnld(self):
ftp = FTP(self.NAS)
ftp.login(user=self.uName, passwd=self.pWord)
for filename in self.dcmfnarr:
remotefn = self.PATH + filename['filepath']
localfn = self.dntarget + os.path.basename(filename['filepath'])
with open(localfn, 'wb') as fp:
ftp.retrbinary("RETR %s" % remotefn, fp.write)
dicom ファイルを選り分ける
そしてやっと、本題です。
カルテ番号で抽出した dicom ファイルは、整理されることなく一緒くたになっています。
これを、検査日とかカルテ番号で選り分けたい。
def taginfo(self, dcmfn):
ds = pydicom.dcmread(dcmfn,force=True)
ptName = ds.PatientName
karteNo = ds.PatientID
studyDate = ds.StudyDate
studyTime = ds.StudyTime
modality = ds.Modality
path = Path(self.localdcm)/Path(f"{karteNo}_{ptName}")/Path(f"{studyDate}_{modality}")
if not os.path.isdir(path):
os.makedirs(path)
copyto = path / os.path.basename(dcmfn)
shutil.move(dcmfn, copyto)
このようにすると、以下のような感じで選り分けされます。
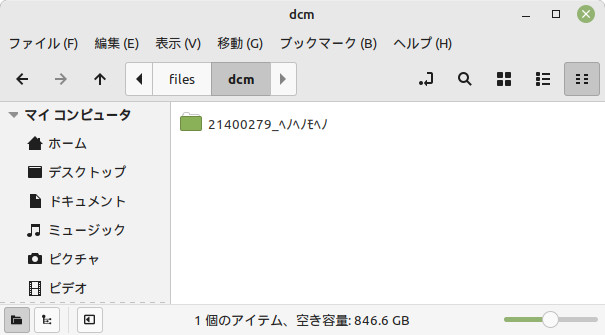
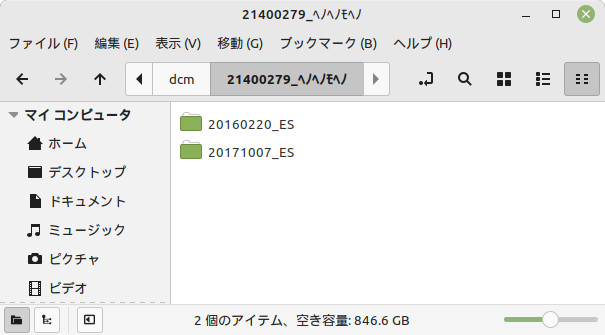
ポイントは、
path = Path(self.localdcm)/Path(f"{karteNo}_{ptName}")/Path(f"{studyDate}_{modality}")
で、この path を変えることによって選り分けの構造をいくらでも変えることができます。
zip ファイルにして端末にダウンロードできるようにする
この病院内に保存されている dicom ファイルをCDRに焼いて患者さんに渡すことはよくありますが、上のように選り分けしてから zip ファイルで端末にダウンロードできるようにすれば便利です。
以下のようにすればできます。
def makezip(self):
shutil.rmtree(self.localtmp)
shutil.make_archive('./tmp/files', format='zip', root_dir='./files')
これを flask から呼び出します。
@app.route("/downloadzip", methods=['POST'])
def downloadzip():
fdl.makezip()
response = make_response()
response.data = open('./tmp/files.zip', "rb").read()
response.headers['Content-Type'] = 'application/octet-stream'
response.headers['Content-Disposition'] = 'attachment; filename=files.zip'
return response

